전체 코드
install.packages("HSAUR2")
library(HSAUR2)
data(heptathlon)
head(heptathlon)
heptathlon
summary(heptathlon)
write.csv(heptathlon, file="/Users/DataAnalytics/MultivariateAnalysis/mva/heptathlon.csv")
# R 2.3
heptathlon$hurdles = max(heptathlon$hurdles) - heptathlon$hurdles
heptathlon$run200m = max(heptathlon$run200m) - heptathlon$run200m
heptathlon$run800m = max(heptathlon$run800m) - heptathlon$run800m
head(heptathlon)
round(cor(heptathlon), 2)
plot(heptathlon, pch=19)
# R 2.4
hep_data = heptathlon[, -8]
head(hep_data)
# Principal component analysis using principal ( using eigen)
hep_pca = princomp(hep_data, cor=T, scores=T)
names(hep_pca)
hep_pca
# Principal component analysis using prcomp (using SVD)
hep_pca2 = prcomp(hep_data, scale=TRUE)
hep_pca2
# R 2.5
summary(hep_pca)
eig_val = hep_pca$sdev^2
eig_val
summary(hep_pca2)
hep_var
# R 2.6
screeplot(hep_pca, type="lines", pch=19, main="Scree plot")
hep_var = hep_pca$sdev^2
hep_var
hep_var_ratio = hep_var/sum(hep_var)
round(hep_var_ratio, 3)
screeplot(hep_pca2, type="lines", pch=19, main="Scree plot")
plot(cumsum(hep_var_ratio), type='b', pch=19, xlab='Component',
ylab='Cumulative Proportion')
title('Variance Explained')
# 주성분계수
hep_pca$loadings[, c(1:2)]
# hep_pca2$rotation[, c(1:2)]
# R 2.7
hep_pca$scores[, c(1:2)]
biplot(hep_pca, cex=0.7, col=c("Red", "Blue"))
title("Biplot")
# hep_pca2$x[c(1:5),c(1:2)]
# biplot(hep_pca2)
[주성분 수 결정 방법]
1. 총변이에 대한 공헌도를 확인한다.(고유값의 누적비율)
- 보유된 주성분들이 다변량 총변이에 대해 주어진 일정 비율 이상을 설명할 수 있기 위함이며, 필요한 최소 개수의 주성분을 보유하기 위해 사용한다.
- 일반적으로 누적정보가 전체정보의 정도를 확보할 수 있다면 나머지 주성분들은 무시하게 되나 상황에 따라 주관적으로 판단하는 기준이다.
> # Principal component analysis using prcomp (using SVD)
> hep_pca2 = prcomp(hep_data, scale=TRUE)
> hep_pca2
Standard deviations (1, .., p=7):
[1] 2.1119364 1.0928497 0.7218131 0.6761411 0.4952441 0.2701029 0.2213617
Rotation (n x k) = (7 x 7):
PC1 PC2 PC3 PC4 PC5 PC6
hurdles -0.4528710 0.15792058 -0.04514996 0.02653873 -0.09494792 -0.78334101
highjump -0.3771992 0.24807386 -0.36777902 0.67999172 0.01879888 0.09939981
shot -0.3630725 -0.28940743 0.67618919 0.12431725 0.51165201 -0.05085983
run200m -0.4078950 -0.26038545 0.08359211 -0.36106580 -0.64983404 0.02495639
longjump -0.4562318 0.05587394 0.13931653 0.11129249 -0.18429810 0.59020972
javelin -0.0754090 -0.84169212 -0.47156016 0.12079924 0.13510669 -0.02724076
run800m -0.3749594 0.22448984 -0.39585671 -0.60341130 0.50432116 0.15555520
PC7
hurdles 0.38024707
highjump -0.43393114
shot -0.21762491
run200m -0.45338483
longjump 0.61206388
javelin 0.17294667
run800m -0.09830963
> summary(hep_pca)
Importance of components:
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6
Standard deviation 2.1119364 1.0928497 0.72181309 0.67614113 0.49524412 0.27010291
Proportion of Variance 0.6371822 0.1706172 0.07443059 0.06530955 0.03503811 0.01042223
Cumulative Proportion 0.6371822 0.8077994 0.88222998 0.94753952 0.98257763 0.99299986
Comp.7
Standard deviation 0.221361710
Proportion of Variance 0.007000144
Cumulative Proportion 1.000000000위의 summary(hep_pca)를 보면 Comp.2의 Cumulative Proportion의 값이 누적 0.8077994로 되어있는 것을 확인할 수 있다.
두 번째 것을 보면 80%를 설명하겠구나, 세 번째 것을 보면 88%를 설명하겠구나 라고 판단할 수 있다.
몇번째 것까지 선택할지는 상황에 따라 주관적으로 선택하면 된다.
2. 개별 고유값의 크기 확인
- 공분산 행렬을 바탕으로 고유값을 구하는데 공분산을 각각의 표준편차로 다시 나눠주면 상관계수가 된다. 각각 나눠서 단위를 똑같이 맞춰주는 것이 상관계수인데 해당 값으로 주성분을 구했다면 상관행렬의 대각원소가 1이므로 각 주성분의 분산은 1이다.
- 1보다 작은 고유값을 가지는 주성분은 원래 변수중의 어느것보다 작은 변이(정보)를 가지므로 고려할 가치가 없다.
- 주성분으로 고려되기 위해서는 그의 분산인 고유값이 적어도 1 이상이 되어야 한다는 ‘Kaiser의 규칙’을 기준으로 사용한다.
- Jolliffe(1972)는 수많은 사례연구를 바탕으로 Kaiser의 고유값이 1이상이어야 한다는 기준 대신 0.7을 제안했다.
> eig_val = hep_pca$sdev^2
> eig_val
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
4.46027516 1.19432056 0.52101413 0.45716683 0.24526674 0.07295558 0.04900101
3. 스크리 그림(Scree diagram)
Cattell(1966): “가파르게(steep)” -> “완만 한(shallow)” 으로 변화하는 지점(커브의 팔꿈치)까지의 주성분 포함한다.
- Scree 그림은 수평축에 주성분 번호를 놓고 수직축에 해당 주성분에 대응하는 고유값을 연결한 그림이다.
- Scree 그림의 어느 구간에서 꺽은 선의 기울기가 가파르게 떨어진 후 완만한 감소를 보인다면, 그 하강지점에 대응되는 개수의 주성분을 보유하는 것이 바람직하다.
- 각 주성분의 분산인 고유값 그 자체보다 내림차순의 인접 고유값들이 가지는 감소의 정도를 통해 적정수준의 주성분을 보유하는 방법이다.
- ‘scree’는 화산폭발에서 산자락에 널려있는 자갈더미를 의미한다.
[주성분 분석의 절차]
1. 자료 가져오기, 요약통계량
heptathlon이라는 자료는 1988년도에 서울 올림픽 육상 여성 7종 경기의 결과다.
> install.packages("HSAUR2")
> library(HSAUR2)
> data(heptathlon)
> head(heptathlon)
hurdles highjump shot run200m longjump javelin run800m score
Joyner-Kersee (USA) 12.69 1.86 15.80 22.56 7.27 45.66 128.51 7291
John (GDR) 12.85 1.80 16.23 23.65 6.71 42.56 126.12 6897
Behmer (GDR) 13.20 1.83 14.20 23.10 6.68 44.54 124.20 6858
Sablovskaite (URS) 13.61 1.80 15.23 23.92 6.25 42.78 132.24 6540
Choubenkova (URS) 13.51 1.74 14.76 23.93 6.32 47.46 127.90 6540
Schulz (GDR) 13.75 1.83 13.50 24.65 6.33 42.82 125.79 6411
> summary(heptathlon)
hurdles highjump shot run200m longjump
Min. :12.69 Min. :1.500 Min. :10.00 Min. :22.56 Min. :4.880
1st Qu.:13.47 1st Qu.:1.770 1st Qu.:12.32 1st Qu.:23.92 1st Qu.:6.050
Median :13.75 Median :1.800 Median :12.88 Median :24.83 Median :6.250
Mean :13.84 Mean :1.782 Mean :13.12 Mean :24.65 Mean :6.152
3rd Qu.:14.07 3rd Qu.:1.830 3rd Qu.:14.20 3rd Qu.:25.23 3rd Qu.:6.370
Max. :16.42 Max. :1.860 Max. :16.23 Max. :26.61 Max. :7.270
javelin run800m score
Min. :35.68 Min. :124.2 Min. :4566
1st Qu.:39.06 1st Qu.:132.2 1st Qu.:5746
Median :40.28 Median :134.7 Median :6137
Mean :41.48 Mean :136.1 Mean :6091
3rd Qu.:44.54 3rd Qu.:138.5 3rd Qu.:6351
Max. :47.50 Max. :163.4 Max. :72912. 자료 변형하기
분석하기 전에 경기 특성에 따라 달리기 같은 종목은 값이 작을수록 좋고 던지기 같은 종목은 값이 클수록 좋다.
이 차이를 일관되게 하기 위해서 값을 변형을 하는 것이 필요하다.
> heptathlon$hurdles = max(heptathlon$hurdles) - heptathlon$hurdles
> heptathlon$run200m = max(heptathlon$run200m) - heptathlon$run200m
> heptathlon$run800m = max(heptathlon$run800m) - heptathlon$run800m
> head(heptathlon)
hurdles highjump shot run200m longjump javelin run800m score
Joyner-Kersee (USA) 3.73 1.86 15.80 4.05 7.27 45.66 34.92 7291
John (GDR) 3.57 1.80 16.23 2.96 6.71 42.56 37.31 6897
Behmer (GDR) 3.22 1.83 14.20 3.51 6.68 44.54 39.23 6858
Sablovskaite (URS) 2.81 1.80 15.23 2.69 6.25 42.78 31.19 6540
Choubenkova (URS) 2.91 1.74 14.76 2.68 6.32 47.46 35.53 6540
Schulz (GDR) 2.67 1.83 13.50 1.96 6.33 42.82 37.64 64113. 상관계수행렬, 산점도행렬 확인
상자그림 분포를 알기 위해 산점도를 함께 그려서 산점도 행렬을 확인하는 것이 좋다.
롱점프와 같이 우상향 하는 행렬들은 서로 선형 상관관계가 있다고 보여진다.
창던지기 부분이 약간 선형성이 떨어진다.
> round(cor(heptathlon), 2)
hurdles highjump shot run200m longjump javelin run800m score
hurdles 1.00 0.81 0.65 0.77 0.91 0.01 0.78 0.92
highjump 0.81 1.00 0.44 0.49 0.78 0.00 0.59 0.77
shot 0.65 0.44 1.00 0.68 0.74 0.27 0.42 0.80
run200m 0.77 0.49 0.68 1.00 0.82 0.33 0.62 0.86
longjump 0.91 0.78 0.74 0.82 1.00 0.07 0.70 0.95
javelin 0.01 0.00 0.27 0.33 0.07 1.00 -0.02 0.25
run800m 0.78 0.59 0.42 0.62 0.70 -0.02 1.00 0.77
score 0.92 0.77 0.80 0.86 0.95 0.25 0.77 1.00
> plot(heptathlon, pch=19)
4. 주성분분석 실행하기
princomp() 함수는 주성분 분석을 실행한다.
4-1) princomp(고유값)
- cor=T 는 주성분 분석을 상관계수로 하겠다는 의미다.
- scores=T 는 주성분분석을 한 뒤에 각각의 케이스가 갖는 주성분 점수들을 구한다는 의미다.
위 인자들을 hep_pca에 넣는다.
names는 결과가 리스트로 들어오니까 리스트로 들어온 이름이 뭔지 확인 하는 것이다.
> # R 2.4
> hep_data = heptathlon[, -8] #자료에서 변수 score를 뺀 나머지 변수로 주성분 분석을 하기 위해 score를 지운다.
> head(hep_data)
hurdles highjump shot run200m longjump javelin run800m
Joyner-Kersee (USA) 3.73 1.86 15.80 4.05 7.27 45.66 34.92
John (GDR) 3.57 1.80 16.23 2.96 6.71 42.56 37.31
Behmer (GDR) 3.22 1.83 14.20 3.51 6.68 44.54 39.23
Sablovskaite (URS) 2.81 1.80 15.23 2.69 6.25 42.78 31.19
Choubenkova (URS) 2.91 1.74 14.76 2.68 6.32 47.46 35.53
Schulz (GDR) 2.67 1.83 13.50 1.96 6.33 42.82 37.64
> # Principal component analysis using principal ( using eigen)
> hep_pca = princomp(hep_data, cor=T, scores=T)
> names(hep_pca)
[1] "sdev" "loadings" "center" "scale" "n.obs" "scores" "call"
> hep_pca
Call:
princomp(x = hep_data, cor = T, scores = T)
Standard deviations:
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
2.1119364 1.0928497 0.7218131 0.6761411 0.4952441 0.2701029 0.2213617
7 variables and 25 observations.4-2) princomp(SVD:Single Value Decomposition 알고리즘 => 특이값)
SVD 알고리즘을 쓰면 행렬모양이 다양해도 상관없기 때문에 일반적으로 이 방식을 더 많이 쓴다.
Standard deviations를 보면 pc들이 구해진다.
> # Principal component analysis using prcomp (using SVD)
> hep_pca2 = prcomp(hep_data, scale=TRUE)
> hep_pca2
Standard deviations (1, .., p=7):
[1] 2.1119364 1.0928497 0.7218131 0.6761411 0.4952441 0.2701029 0.2213617
Rotation (n x k) = (7 x 7):
PC1 PC2 PC3 PC4 PC5 PC6 PC7
hurdles -0.4528710 0.15792058 -0.04514996 0.02653873 -0.09494792 -0.78334101 0.38024707
highjump -0.3771992 0.24807386 -0.36777902 0.67999172 0.01879888 0.09939981 -0.43393114
shot -0.3630725 -0.28940743 0.67618919 0.12431725 0.51165201 -0.05085983 -0.21762491
run200m -0.4078950 -0.26038545 0.08359211 -0.36106580 -0.64983404 0.02495639 -0.45338483
longjump -0.4562318 0.05587394 0.13931653 0.11129249 -0.18429810 0.59020972 0.61206388
javelin -0.0754090 -0.84169212 -0.47156016 0.12079924 0.13510669 -0.02724076 0.17294667
run800m -0.3749594 0.22448984 -0.39585671 -0.60341130 0.50432116 0.15555520 -0.098309635. 주성분 분석 결과: 요약, 고유값
- 결과로 고유값을 구하고 주성분 돌린 결과로 summary 해보면 우리가 꼭 봐야하는 Cumulative Proportion 보여준다.
- 두 번째 주성분인 Comp.2 의 Cumulative Proportion을 보면 0.8077994이라서 두 번째 주성분까지 채택을 하면 약 80%의 설명력을 얻을 수 있다.
- eig_val 할 때 hep_pca$sdev^2 제곱값으로 분산의 크기를 확인한다.
- comp.2는 1 이상이어서 포함시키고 comp.3은 0.7 미만아어서 탈락시킨다.
> summary(hep_pca)
Importance of components:
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
Standard deviation 2.1119364 1.0928497 0.72181309 0.67614113 0.49524412 0.27010291 0.221361710
Proportion of Variance 0.6371822 0.1706172 0.07443059 0.06530955 0.03503811 0.01042223 0.007000144
Cumulative Proportion 0.6371822 0.8077994 0.88222998 0.94753952 0.98257763 0.99299986 1.000000000
> eig_val = hep_pca$sdev^2
> eig_val
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
4.46027516 1.19432056 0.52101413 0.45716683 0.24526674 0.07295558 0.04900101
> summary(hep_pca2)
Importance of components:
PC1 PC2 PC3 PC4 PC5 PC6 PC7
Standard deviation 2.1119 1.0928 0.72181 0.67614 0.49524 0.27010 0.2214
Proportion of Variance 0.6372 0.1706 0.07443 0.06531 0.03504 0.01042 0.0070
Cumulative Proportion 0.6372 0.8078 0.88223 0.94754 0.98258 0.99300 1.0000
> hep_var
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
4.46027516 1.19432056 0.52101413 0.45716683 0.24526674 0.07295558 0.049001016. 스크리그림, 누적분산
첫 번째, 두 번째가 완전 가파르다가 세 번째부터 완만해진다. 두 번째 까지는 선택했을 경우 큰 영향이 있지만 세 번째 것은 도움이 될지 잘 판단하여 선택해야 한다.
이런식으로 표로 보면 직관적으로 확인할 수 있어서 좋다.
> # 스크리 그림
> screeplot(hep_pca, type="lines", pch=19, main="Scree plot")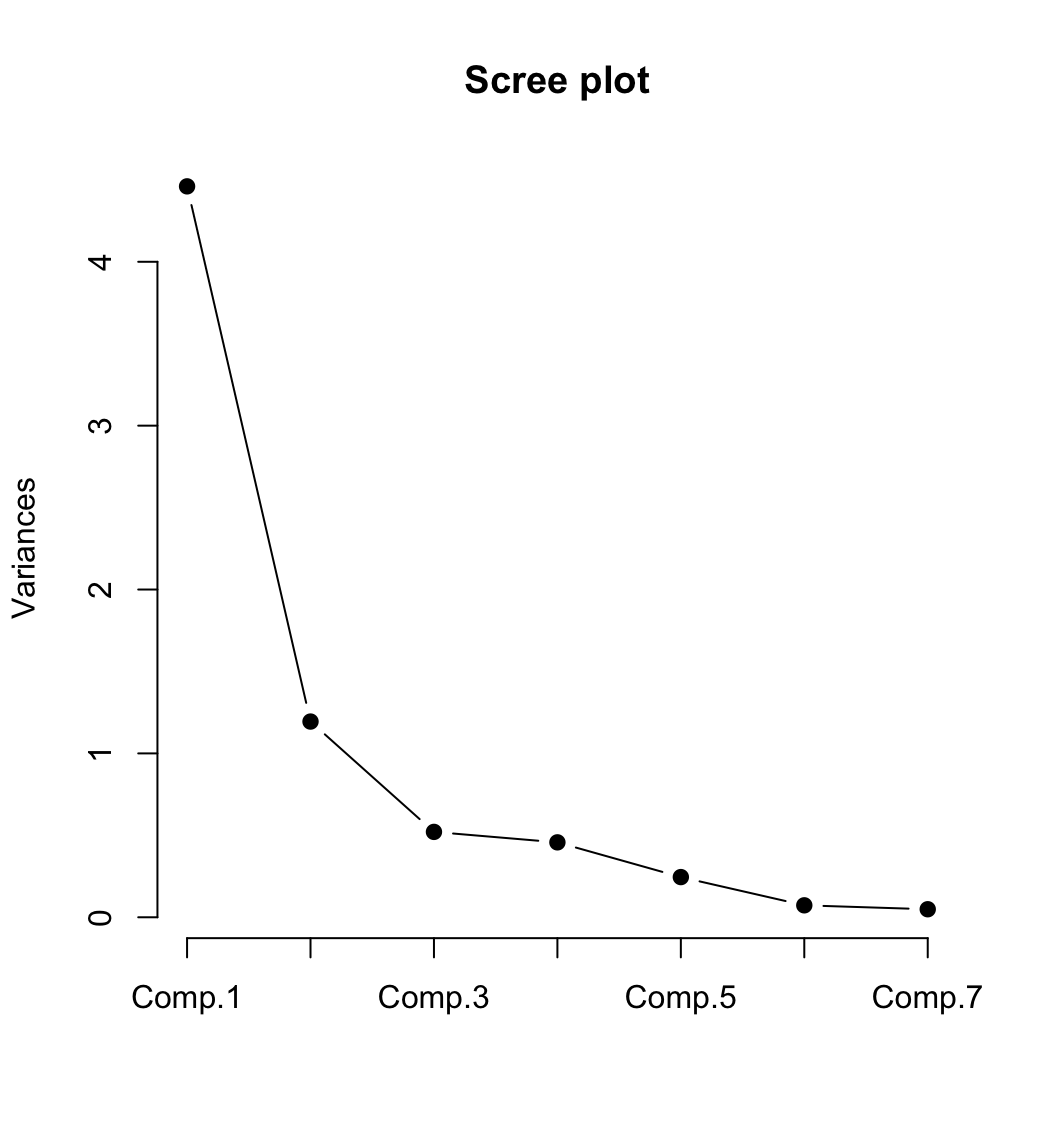
> hep_var = hep_pca$sdev^2
> hep_var
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
4.46027516 1.19432056 0.52101413 0.45716683 0.24526674 0.07295558 0.04900101
> hep_var_ratio = hep_var/sum(hep_var)
> round(hep_var_ratio, 3)
Comp.1 Comp.2 Comp.3 Comp.4 Comp.5 Comp.6 Comp.7
0.637 0.171 0.074 0.065 0.035 0.010 0.007
> plot(cumsum(hep_var_ratio), type='b', pch=19, xlab='Component',
+ ylab='Cumulative Proportion')
7. 주성분계수
- 주성분 계수 구하고 loadings 찾으면 된다.
- 소수점 3째자리까지 보여주고 가중치로 쓰는 곳도 있다.
> # 주성분계수
> hep_pca$loadings[, c(1:2)]
Comp.1 Comp.2
hurdles 0.4528710 0.15792058
highjump 0.3771992 0.24807386
shot 0.3630725 -0.28940743
run200m 0.4078950 -0.26038545
longjump 0.4562318 0.05587394
javelin 0.0754090 -0.84169212
run800m 0.3749594 0.22448984
> # hep_pca2$rotation[, c(1:2)]첫 번째 주성분은 모든 변수의 절대값이 큰 값을 가지는 것으로 보아 전반적인 체력정도를 나타내는 성분으로 판단할 수 있다.
두 번째 주성분은 창던지기의 계수가 다른 변수에 비해 상대적으로 절대값이 큰 것을 보아 창던지기와 밀접한 관련이 있는 것으로 판단할 수 있다.
8. 주성분점수, 행렬도
- PC1, PC2에 따른 주성분 점수 구하고 행렬도 구하는 순서다.
- 1부터 2까지 선택해서 봤을 때 아래처럼 나온다.
> # R 2.7
> hep_pca$scores[, c(1:2)]
Comp.1 Comp.2
Joyner-Kersee (USA) 4.20643487 -1.26802363
John (GDR) 2.94161870 -0.53452561
Behmer (GDR) 2.70427114 -0.69275901
Sablovskaite (URS) 1.37105209 -0.70655862
Choubenkova (URS) 1.38704979 -1.78931718
Schulz (GDR) 1.06537236 0.08104469
Fleming (AUS) 1.12307639 0.33042906
Greiner (USA) 0.94221015 0.82345074
Lajbnerova (CZE) 0.54118484 -0.14933917
Bouraga (URS) 0.77548704 0.53686251
Wijnsma (HOL) 0.56773896 1.42507414
Dimitrova (BUL) 1.21091937 0.36106077
Scheider (SWI) -0.01578005 -0.82307249
Braun (FRG) -0.00385205 -0.72953750
Ruotsalainen (FIN) -0.09261899 -0.77877955
Yuping (CHN) 0.14005513 0.54831883
Hagger (GB) -0.17465745 1.77914066
Brown (USA) -0.52996001 -0.74195530
Mulliner (GB) -1.14869009 0.64788023
Hautenauve (BEL) -1.10808552 1.88531477
Kytola (FIN) -1.47689483 0.94353198
Geremias (BRA) -2.05556037 0.09495979
Hui-Ing (TAI) -2.93969248 0.67514662
Jeong-Mi (KOR) -3.03136461 0.97939889
Launa (PNG) -6.39931438 -2.89774561
> biplot(hep_pca, cex=0.7, col=c("Red", "Blue"))
> title("Biplot")
biplot 행렬도를 보면 두개를 동시에 보여준다.
아래는 component1에 대한 것이고 X축은 component2에 대한 것이다.
파란색은 주성분계숨이며 위치와 벡터의 방향에 따라 해석을 하게 된다.
biplot에서 파란색 아래에쪽으로 난 화살표는 창던지기인데 혼자 아래로 튀어나와 있고 다른 것들은 오른쪽 화살표로 몰려있는 것을 보면 3시~4시 방향에 있는 화살표들이 유사한 성향을 나타낸다고 볼 수 있다.
가까이 있는 것일수록 상관성이 높다.
주성분계수 마이너스 부호는 신경쓸 필요 없다(절대값으로 확인)
Python
# Py 2.1
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
# 데이터 읽기
heptathlon = pd.read_csv("/Users/DataAnalytics/MultivariateAnalysis/mva/heptathlon.csv")
# heptathlon = pd.read_csv("/Users/DataAnalytics/MultivariateAnalysis/mva/heptathlon.csv")
heptathlon.head(3)
# 변수이름 확인하기
heptathlon.columns
# 기술통계량 구하기 – 소수점 이하 2자리 반올림 표시
round(heptathlon.describe(), 2)
#Py 2.2
# 변환: 변수최댓값 - 변숫값
heptathlon.hurdles = np.max(heptathlon.hurdles) - heptathlon.hurdles
heptathlon.run200m = np.max(heptathlon.run200m) - heptathlon.run200m
heptathlon.run800m = np.max(heptathlon.run800m) - heptathlon.run800m
heptathlon.head()
# 분석변수 선택하기
feature = ['hurdles','highjump','shot','run200m','longjump','javelin','run800m']
hep_data = heptathlon[feature]
# hep_data = heptathlon.iloc{:, 1:8]
# hep_data = heptathlon.iloc{:, 1:-1]
# 변수 표준화
from sklearn.preprocessing import StandardScaler
x = StandardScaler().fit_transform(hep_data)
# Py 2.3
# 초기 주성분분석
from sklearn.decomposition import PCA
pca_init = PCA(n_components=len(hep_data.columns))
pca_init.fit(x)
pca_init.explained_variance_
np.cumsum(pca_init.explained_variance_ratio_)
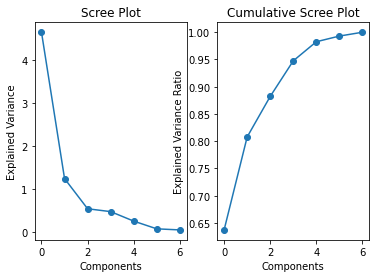
# 스크리 그림 그리기
plt.figure()
plt.subplot(121)
plt.plot(pca_init.explained_variance_, 'o-')
plt.title('Scree Plot')
plt.xlabel('Components')
plt.ylabel('Explained Variance')
plt.subplot(122)
plt.plot(np.cumsum(pca_init.explained_variance_ratio_), 'o-')
plt.title('Cumulative Scree Plot')
plt.xlabel('Components')
plt.ylabel('Explained Variance Ratio')
plt.show()
# Py 2.4
# 주성분분석 – 주성분 수 2개 추출
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
hep_pca = pca.fit_transform(x)
# dir(pca)
# 주성분분산
pca.explained_variance_
# 주성분분산 비율
pca.explained_variance_ratio_
# 주성분계수
np.round(pca.components_, 3)
# 주성분점수
hep_pca[0:5,:]
# Py 2.5
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
# 데이터 읽기
beer = pd.read_csv("/beer.csv")
# beer = pd.read_csv("/Users/DataAnalytics/MultivariateAnalysis/mva/beer.csv")
beer.head()
# 기술통계량 구하기
beer.describe()
# Py 2.6
# 주성분분석 – 주성분 수 3으로 함
from sklearn.decomposition import PCA
pca = PCA(n_components=3)
pca_beer = pca.fit_transform(beer)
# 주성분분산
pca.explained_variance_
# 주성분 표준편차
np.sqrt(pca.explained_variance_)
# 주성분분산 비율
pca.explained_variance_ratio_
# 주성분계수
np.round(pca.components_, 3)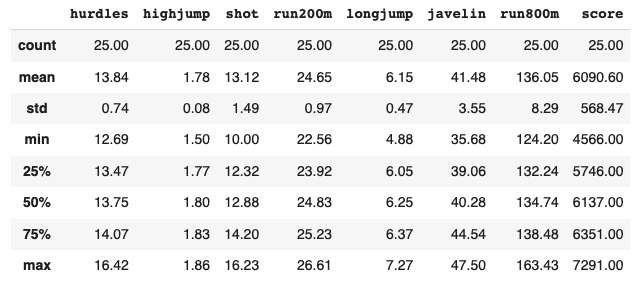



'Data Science > Multivariate Analysis' 카테고리의 다른 글
| [Multivariate Analysis] Factor analysis(인자분석) (0) | 2022.03.21 |
|---|---|
| [Multivariate Analysis] Principal Component Analysis(PCA) 주성분분석 (0) | 2022.03.18 |
| [Mulivariate Analysis] eigenvalue(고유값)과 eigenvector(고유벡터) (0) | 2022.03.18 |
| [Multivariate Analysis] 다차원 통계그래프 (0) | 2022.03.13 |
| [Multivariate Analysis] 이변량 통계그래프 (0) | 2022.03.13 |




댓글